Innovazione Biotech e Diagnostica
Real-Time PCR decentralizzata per patologie respiratorie
Diagnostica molecolare centralizzata e distribuita: differenze, vantaggi e integrazione dei modelli in ottica post-pandemica
Abstract
L’adozione di piattaforme Real-Time PCR multiplex decentralizzate rappresenta un cambiamento di paradigma nella diagnosi delle infezioni respiratorie acute. Queste soluzioni, spesso costituite da dispositivi portatili connessi a sistemi cloud, consentono di identificare simultaneamente influenza A/B, virus respiratorio sinciziale (RSV), SARS-CoV-2 e altri patogeni (inclusi marker di coinfezioni batteriche) direttamente presso il punto di cura. Ciò riduce drasticamente il Turn-Around Time (TAT) diagnostico – passando da giorni a ore – e migliora la gestione clinica: i medici possono isolare subito i pazienti contagiosi, avviare terapie antivirali mirate entro poche ore dal ricovero e evitare antibiotici inutili nei casi virali[1][2]. Nel complesso, la Real-time PCR decentralizzata combina la sensibilità elevata dei test di laboratorio con la praticità del point-of-care, offrendo un potente strumento per rafforzare la stewardship antibiotica e prendere decisioni critiche in tempi record, sia in ospedale che in contesti periferici. Di seguito, esaminiamo la struttura tecnologica di queste soluzioni, le patologie coperte, i limiti degli approcci antigenici tradizionali e i benefici concreti apportati da piattaforme come bCUBE™ e bAPP™. Un approfondimento è dedicato ai casi d’uso e all’impatto clinico, con la testimonianza diretta del CTO di Ulisse Biomed, Lorenzo Colombo.
- Snapshot
- Introduzione
- 1. Struttura tecnologica e modello decentralizzato
- 2. Patologie coperte e coinfezioni rilevabili
- 3. Limiti degli approcci antigenici rapidi
- 4. Il valore aggiunto della PCR in tempo reale multiplex
- 5. bCUBE™ e bAPP™: un caso di innovazione italiana
- 6. Casi d’uso in corsia e sul territorio
- 7. Impatto clinico e operativo
- Conclusioni
Snapshot
- Ct (Cycle Threshold): parametro quantitativo della PCR in tempo reale. Corrisponde al numero di cicli necessari perché il segnale di amplificazione superi il livello di soglia; valori di Ct più bassi indicano cariche virali più elevate nel campione.
- TAT (Turn-Around Time): tempo di ciclo totale tra il prelievo del campione e la refertazione del risultato. Nella diagnostica tradizionale di laboratorio il TAT per un test molecolare respiratorio può essere di molte ore; con soluzioni decentralizzate può scendere sotto 1 ora.
- qPCR (PCR in tempo reale quantitativa): tecnica di Polymerase Chain Reaction che consente di monitorare in tempo reale l’amplificazione del materiale genetico, mediante sonde fluorescenti. È definita “quantitativa” perché permette anche di stimare la quantità iniziale di target (virus, batterio) nel campione, grazie al valore di Ct.
- Multiplex (PCR multiplex): approccio che prevede l’amplificazione simultanea di più bersagli molecolari in un’unica reazione PCR. Si ottiene utilizzando differenti coppie di primer/sonde marcate con fluorofori diversi. Nel contesto dei test respiratori, il multiplex consente di rilevare più virus (es. influenza, RSV, SARS-CoV-2) in parallelo dallo stesso campione.
- Coinfezione: presenza contemporanea di più di un agente infettivo nello stesso paziente. Nel caso delle infezioni respiratorie, può trattarsi di coinfezione virale (es. influenza + RSV) o virus-batterio (es. influenza che predispone a polmonite da pneumococco). Le coinfezioni possono aggravare il quadro clinico e richiedono terapie combinate; identificarle è importante per la gestione ottimale.
Introduzione
Nel contesto delle infezioni respiratorie acute – dall’influenza stagionale alle polmoniti virali come COVID-19 – la rapidità e l’accuratezza della diagnosi giocano un ruolo fondamentale. Riconoscere subito l’agente patogeno significa poter isolare i pazienti contagiosi, iniziare precocemente trattamenti specifici (ad esempio antivirali per l’influenza) ed evitare terapie inutili. Purtroppo, i metodi tradizionali di diagnosi di laboratorio (PCR centralizzata o colture) richiedono spesso diverse ore o giorni per fornire un risultato, ritardando le decisioni cliniche cruciali[1]. Durante la pandemia di COVID-19 si è evidenziato come un approccio solo centralizzato alla diagnostica possa creare colli di bottiglia logistici e tempi di attesa insostenibili. Questo ha accelerato lo sviluppo di test point-of-care molecolari: strumenti portatili capaci di eseguire analisi di biologia molecolare direttamente sul luogo di cura (ambulatorio, pronto soccorso, reparto) con prestazioni paragonabili al laboratorio. In particolare, le piattaforme di Real-Time PCR decentralizzata in modalità multiplex stanno emergendo come game-changer nella diagnostica respiratoria. Queste soluzioni combinano la gold standard PCR (riconosciuta come metodica di riferimento per sensibilità e specificità[3]) con la flessibilità di dispositivi compatti e connessi. Il risultato è una rete di “mini-laboratori” distribuiti che consente referti entro un paio d’ore, rivoluzionando la gestione di patologie come influenza A/B, RSV, SARS-CoV-2 e le loro possibili coinfezioni. Nei paragrafi seguenti analizzeremo in dettaglio come funziona questa tecnologia, quali vantaggi offre rispetto ai test antigenici rapidi e qual è l’impatto operativo nei contesti clinici.
1. Struttura tecnologica e modello decentralizzato
Le soluzioni di Real-time PCR decentralizzate si basano su strumentazione portatile e connessa in rete, combinata con software cloud per il controllo e l’analisi dei dati. Un esempio rappresentativo è la piattaforma Hyris di Ulisse Biomed, composta dal dispositivo bCUBE™ e dall’applicativo cloud bAPP™. bCUBE™ è un termociclatore miniaturizzato, grande circa quanto il palmo di una mano (10x10x12 cm, ~1 kg), ma in grado di eseguire amplificazioni geniche con performance da laboratorio [4]. Nonostante le dimensioni ridotte, questo device supporta cicli termici rapidi e controllati e può integrare fino a 4 canali ottici, permettendo analisi multiplex ad ampio spettro (ad esempio, rilevare contemporaneamente più virus). I campioni sono alloggiati in cartucce monouso da 16 o 36 pozzetti, e più unità bCUBE possono operare in parallelo aumentando linearmente il throughput [5]. L’installazione è plug-and-play: basta l’alimentazione elettrica e una connessione (Ethernet o Wi-Fi) per iniziare a operare, senza necessità di calibrazioni complesse o infrastrutture di laboratorio [6].
Il cervello del sistema risiede in bAPP™, una piattaforma software cloud che consente di controllare da remoto uno o più dispositivi e accedere ai dati in tempo reale [7]. Tramite un’interfaccia web intuitiva, l’operatore può avviare protocolli PCR preimpostati o personalizzati, monitorare in diretta le curve di amplificazione e ricevere dal sistema un’interpretazione automatica del risultato. Quest’ultimo aspetto è cruciale: gli algoritmi di machine learning/AI integrati supportano l’analisi di output complessi, ad esempio riconoscendo la presenza di patogeni multipli da curve fluorescenti multi-canale [8]. In pratica, il software può fornire un referto immediato (es. “positivo per Influenza A e RSV”), riducendo la dipendenza da personale altamente specializzato per interpretare i dati grezzi. Tutti i risultati sono salvati nel database cloud e accessibili in tempo reale da qualunque sede: ciò abilita modelli operativi innovativi, come la supervisione a distanza da parte di un laboratorio centrale o l’integrazione con i sistemi informativi ospedalieri (LIS/LIMS) per una refertazione unificata. Il modello che emerge è quello di una diagnostica “cloud-based” distribuita: dispositivi molecolari compatti dislocati sul territorio o nei reparti, orchestrati da una piattaforma centrale intelligente.
2. Patologie coperte e coinfezioni rilevabili
La sindrome influenzale e le altre infezioni respiratorie acute possono essere causate da una molteplicità di virus e talvolta da coinfezioni virus-virus o virus-batteri. I sintomi clinici (febbre, tosse, dispnea, mal di gola, ecc.) spesso si sovrappongono, rendendo difficile distinguere l’eziologia senza un test di laboratorio. I pannelli multiplex PCR nascono per affrontare proprio questa sfida: con un unico tampone rinofaringeo, è possibile testare contemporaneamente un ampio spettro di patogeni respiratori. Le piattaforme decentralizzate oggi disponibili coprono tipicamente virus influenzali A e B, RSV (respiratory syncytial virus), SARS-CoV-2 e talvolta altri virus comuni (es. adenovirus, rhinovirus/enterovirus, metapneumovirus) a seconda del kit utilizzato. Alcuni pannelli molecolari sindromici includono anche target per patogeni batterici atipici (come Mycoplasma pneumoniae o Bordetella pertussis), oppure geni di resistenza antibiotica, fornendo un quadro diagnostico completo in caso di polmonite. Identificare una coinfezione batterica in un paziente con influenza, ad esempio, può indirizzare verso una terapia combinata antivirale + antibiotico, mentre confermare che un quadro clinico è dovuto unicamente a virus può far risparmiare antibiotici. Studi recenti evidenziano che con l’avvento di test multiplex molecolari la rilevazione di coinfezioni virali/batteriche è aumentata significativamente, specialmente nelle polmoniti comunitarie [9]. Questo ha implicazioni cliniche importanti: riconoscere subito una coinfezione permette di gestire il paziente in modo più appropriato (ad esempio isolandolo per prevenire la trasmissione virale, ma anche somministrando precocemente antibiotici mirati per il patogeno batterico identificato).
In contesti come il pronto soccorso pediatrico, dove RSV e influenza circolano insieme durante la stagione invernale, disporre di un test multiplex rapido consente di individuare doppie infezioni (es. influenza + streptococco) che altrimenti verrebbero facilmente perse con test singoli. Anche durante la pandemia di COVID-19, la presenza simultanea di SARS-CoV-2 e altri patogeni respiratori (influenza in primis) è stata documentata; per questo oggi molti test multiplex “respiratori” includono in un solo pannello sia i virus influenzali che il coronavirus SARS-CoV-2, facilitando la diagnosi differenziale e l’eventuale rilevazione di co-infezioni virali.
3. Limiti degli approcci antigenici rapidi
Prima dell’ampia diffusione della PCR rapida, la diagnostica decentralizzata delle infezioni respiratorie si basava soprattutto su test antigenici rapidi (es. test immunocromatografici per influenza o RSV, e più recentemente test antigenici per COVID-19). Questi saggi offrono risultati in 10-15 minuti e sono semplici da eseguire, ma presentano importanti limiti in sensibilità. Ad esempio, i Rapid Influenza Diagnostic Tests (RIDT), pur essendo utili per uno screening immediato, possono mancare una proporzione significativa di casi positivi. Secondo il CDC, i test antigenici influenzali devono essere interpretati con prudenza: la sensibilità rispetto alla PCR è sub-ottimale, con frequenti falsi negativi soprattutto nei periodi di alta circolazione virale [10] [11]. In altre parole, un risultato antigenico negativo non esclude realmente l’infezione influenzale attiva, e in presenza di forte sospetto clinico il CDC raccomanda comunque di considerare una terapia antivirale o di confermare con un test molecolare [12]. Limiti analoghi valgono per i test rapidi RSV e per molti test antigenici combinati. La specificità dei test antigenici è generalmente buona, ma anche i falsi positivi possono verificarsi (specie per influenza B in alcuni kit e in bassa prevalenza di malattia) [13].
In sintesi, l’approccio antigenico soffre di un trade-off fra velocità e accuratezza: fornisce un responso veloce ma rischia di sottostimare i casi, soprattutto nelle prime fasi dell’infezione (quando la carica virale è al limite di rilevabilità antigenica) o per alcuni virus in particolare. Inoltre, i test antigenici rilevano tipicamente un patogeno per volta; esistono kit combinati (ad esempio tripli test COVID/Influenza/RSV), ma il livello di multiplexing è limitato e comunque ognuno di questi componenti mantiene i vincoli di sensibilità del singolo antigene [14]. La PCR multiplex, invece, offre sia un’elevata sensibilità sia l’ampliamento del numero di target rilevabili in parallelo, unendo il meglio dei due mondi: rapidità e affidabilità diagnostica.
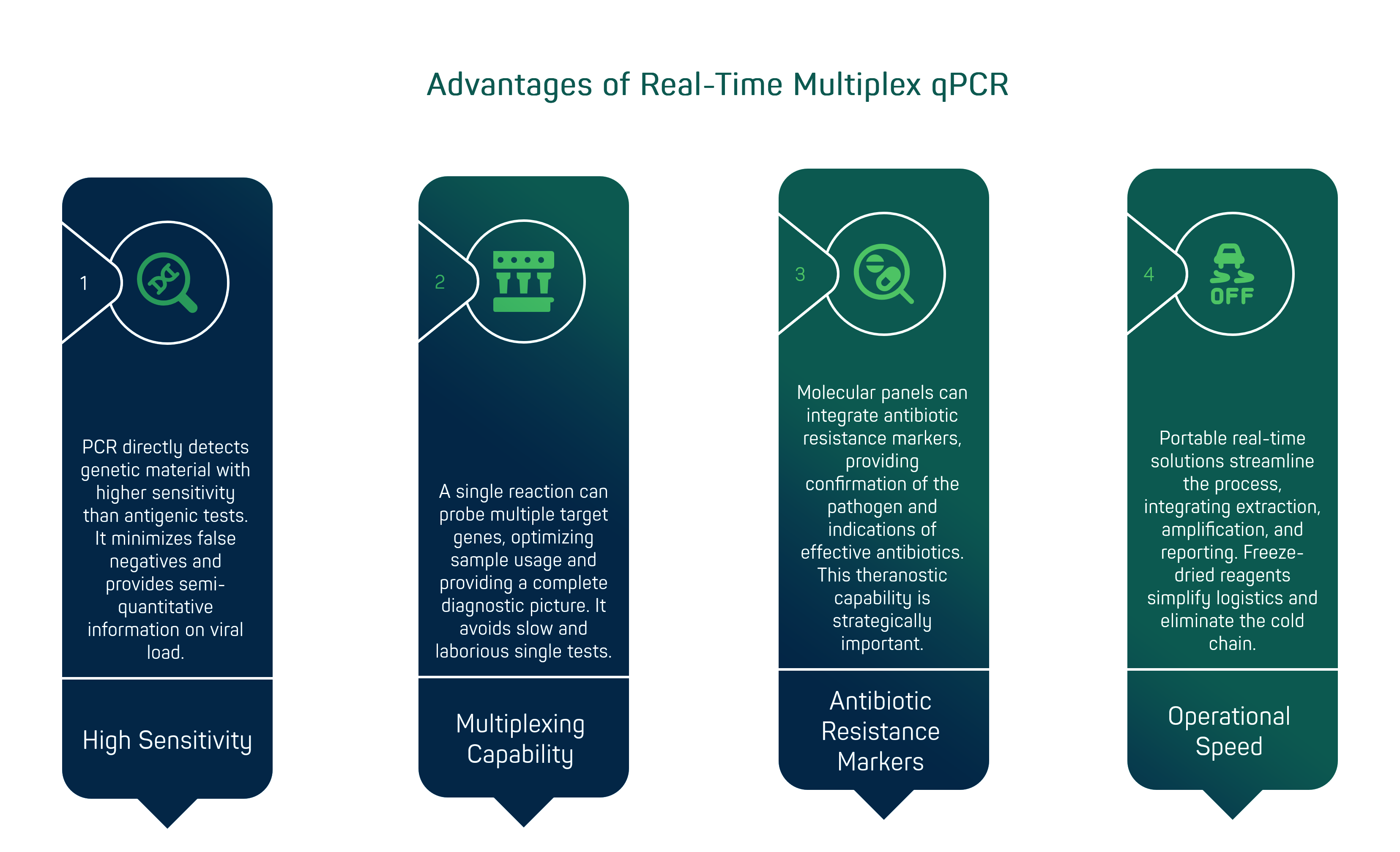
4. Il valore aggiunto della PCR in tempo reale multiplex
La qPCR multiplex in tempo reale presenta diversi vantaggi chiave che la rendono una tecnologia di riferimento per la diagnostica infettivologica avanzata. In primo luogo, la PCR rileva direttamente il materiale genetico (RNA/DNA) del patogeno con una sensibilità molto superiore ai test antigenici, specialmente nelle fasi iniziali dell’infezione o in presenza di bassa carica virale. È noto che i metodi di amplificazione degli acidi nucleici come RT-PCR rappresentano oggi il gold standard per confermare le infezioni virali respiratorie [3]. Ciò si traduce in una capacità di identificare praticamente tutti i casi infetti, riducendo al minimo i falsi negativi. Inoltre, la lettura quantitativa (valori di Ct, cycle threshold) fornisce informazioni semiquantitative sulla carica virale, potenzialmente utili per valutare la fase dell’infezione o la risposta al trattamento.
Il secondo vantaggio è insito nel termine multiplex: una singola reazione può sondare molteplici geni bersaglio. Ad esempio, un pannello respiratorio multiplex può includere primer e sonde per influenza A (gene M), influenza B, RSV, SARS-CoV-2 (gene N e ORF1ab), più eventualmente controlli interni e target batterici. Questo approccio ottimizza il campione – un solo prelievo per tanti test – e offre un quadro diagnostico completo con un unico esame. In situazioni cliniche dove i sintomi sono aspecifici (tosse, febbre) ma la lista dei possibili agenti eziologici è lunga, il multiplex evita la sequenza lenta e laboriosa di test singoli (first influenza, if negative then RSV, if negative then... e così via).
Un ulteriore valore aggiunto è la possibilità di integrare, nei pannelli molecolari, anche marker di antibiotico-resistenza o geni caratteristici di ceppi particolari. Ad esempio, pannelli sindromici di nuova generazione per polmoniti ospedaliere uniscono la ricerca di virus respiratori e batteri (S. pneumoniae, Legionella, ecc.) a quella di geni di resistenza (come mecA per MRSA). In un’unica sessione PCR, il clinico ottiene sia la conferma di quale patogeno sta causando l’infezione, sia indicazioni su quali antibiotici potrebbero (non) essere efficaci. Questa capacità teranostica (diagnosi + indicazione terapeutica) è un elemento di grande rilevanza strategica, specie in un’era di preoccupante resistenza antimicrobica.
Da un punto di vista operativo, la rapidità rimane il punto cardine: a differenza dei protocolli PCR tradizionali in laboratorio (che includono estrazione, amplificazione su termociclatori standard, eventuale invio del referto), le soluzioni real-time portatili integrano il tutto in un flusso snello. Molti dispositivi accettano il campione grezzo (aspirato nasofaringeo, tampone in terreno, ecc.) senza richiedere un’estrazione manuale di RNA, grazie a kit chimici ottimizzati come la linea Sagitta di Ulisse Biomed [15]. Reagenti liofilizzati stabili a temperatura ambiente semplificano la logistica eliminando la catena del freddo, caratteristica critica per operare in ambienti periferici o paesi emergenti [16]. In sintesi, il valore della PCR multiplex decentralizzata sta nell’unire l’affidabilità del laboratorio con la prontezza del point-of-care, abilitando un nuovo standard di diagnosi rapida e completa direttamente al letto del paziente.
5. bCUBE™ e bAPP™: un caso di innovazione italiana
Come accennato, il sistema bCUBE™ + bAPP™ di Ulisse Biomed rappresenta uno dei paradigmi di questa nuova diagnostica decentralizzata. Vale la pena approfondire come queste soluzioni si concretizzino in pratica. bCUBE™ è in sostanza un piccolo laboratorio molecolare portatile. Le sue caratteristiche tecniche assicurano che le prestazioni analitiche siano sovrapponibili a quelle dei termociclatori da banco di un laboratorio centralizzato [17]. Ciò è confermato da studi di validazione: in test comparativi, bCUBE ha dimostrato un’accuratezza analitica pari ai termociclatori standard e piena conformità agli standard di qualità internazionali, risultando certificato secondo l’IVDR europeo e le normative equivalenti nordamericane [18]. In termini di scalabilità, l’architettura modulare permette di aggiungere più unità bCUBE in rete: un ospedale potrebbe distribuire un dispositivo in ogni reparto (es. pronto soccorso, terapia intensiva, reparto infettivi) e farli operare contemporaneamente, connessi alla stessa piattaforma cloud.
Dal canto suo, bAPP™ funge da console di comando e repository centrale. Attraverso bAPP, un responsabile di laboratorio può gestire da remoto decine di analizzatori dislocati geograficamente, vedere in diretta lo stato di ogni test e ricevere alert al completamento dell’analisi. Ad esempio, un laboratorio di microbiologia potrebbe ricevere istantaneamente sul proprio sistema LIS l’esito di un test influenza fatto al pronto soccorso a 2 km di distanza, pochi minuti dopo il triage del paziente. Questo consente di centralizzare la sorveglianza pur decentralizzando l’esecuzione: un modello in cui il test avviene sul campo, ma i dati confluiscono immediatamente a un hub centrale per essere aggregati, monitorati e (se serve) notificati alle autorità sanitarie. Lorenzo Colombo, CTO di Ulisse Biomed, sintetizza così l’approccio: «Abbiamo portato la qualità di un laboratorio centralizzato direttamente sul campo. Con bCUBE e bAPP, il laboratorio segue il paziente ovunque ce ne sia bisogno, mentre i dati viaggiano automaticamente verso il centro per analisi epidemiologiche in tempo reale». Questa filosofia “hub & spoke” invertito permette di mantenere elevati standard analitici e al contempo di democratizzare l’accesso ai test avanzati anche in strutture periferiche o in situazioni dove il tempo è critico (outbreak, emergenze, contesti rurali).
6. Casi d’uso in corsia e sul territorio
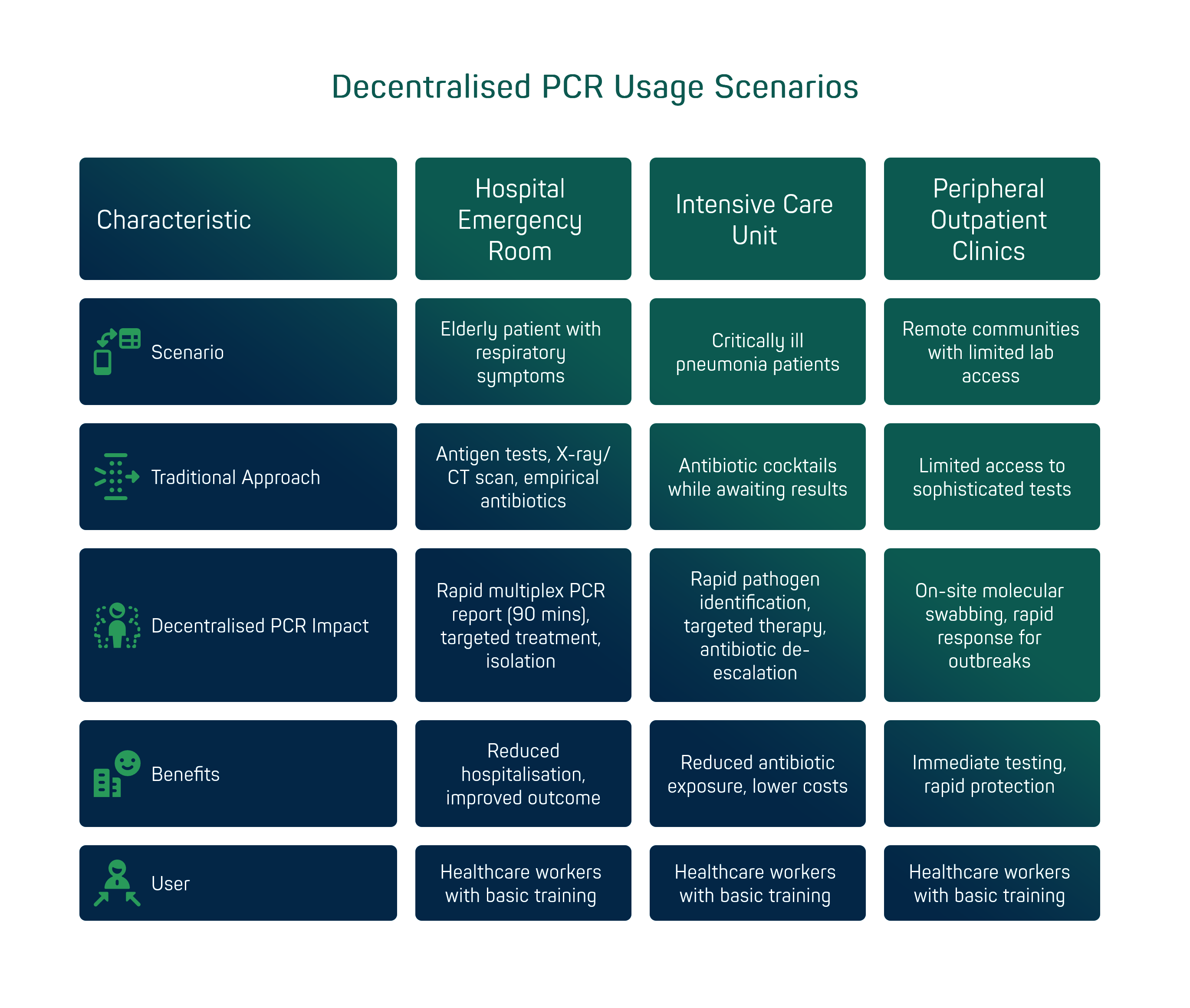
Per comprendere l’impatto pratico della PCR decentralizzata, è utile considerare alcuni scenari d’uso tipici. Pronto soccorso ospedaliero: un paziente anziano arriva con febbre alta, tosse e dispnea in pieno inverno. I sintomi potrebbero essere dovuti a influenza, COVID-19, polmonite batterica o una combinazione di essi. Nella routine tradizionale, si eseguirebbe un test antigenico influenza e COVID (avendo però dubbi sull’affidabilità del risultato negativo) e comunque si avvierebbe una radiografia/TC per sospetta polmonite, somministrando intanto antibiotici empirici. Con un sistema PCR point-of-care multiplex, invece, dal tampone nasofaringeo eseguito all’arrivo si ottiene entro 90 minuti un referto completo: ad esempio “Influenza A e Streptococcus pneumoniae positivi; RSV e SARS-CoV-2 negativi”. Ciò permette in meno di due ore di: isolare il paziente in area influenza (evitando contagi in reparto), iniziare subito l’antivirale specifico (oseltamivir) per l’influenza A e allo stesso tempo somministrare l’antibiotico mirato per lo pneumococco (coinfezione batterica), evitando magari altri antibiotici a largo spettro non necessari. Il risultato? Un management ottimizzato che può ridurre i tempi di degenza e migliorare nettamente l’esito clinico [1].
Reparto di terapia intensiva: pazienti critici con polmonite grave spesso ricevono cocktail di antibiotici in attesa di chiarire se l’origine è batterica, virale o entrambe. L’uso di pannelli PCR ampliati (come quelli per polmoniti nosocomiali) direttamente in reparto consente di avere entro poche ore l’identificazione dei patogeni coinvolti e dei geni di resistenza. Ad esempio, rilevare Staphylococcus aureus con gene mecA negativo potrebbe far sospendere una terapia anti-MRSA inutile, oppure identificare un virus (metapneumovirus) dietro a un quadro respiratorio atipico porterebbe a de-escalation antibiotica immediata. Si tratta di decisioni che tradizionalmente avrebbero atteso 2-3 giorni (colture, risultati centralizzati), mentre con la diagnostica decentralizzata avvengono in giornata, alleggerendo l’esposizione del paziente ad antibiotici inutili e riducendo i costi ospedalieri.
Ambulatori periferici e cliniche territoriali: in comunità remote o semplicemente fuori dai grandi centri urbani, l’accesso ai test di laboratorio sofisticati è spesso limitato. Un sistema portatile come bCUBE può essere installato in un piccolo laboratorio distrettuale o addirittura in una farmacia clinica, abilitando servizi come il tampone molecolare per influenza e COVID con risposta in loco. Ciò è stato particolarmente utile durante i picchi pandemici, ma rimane un modello valido oltre la pandemia: pensiamo alle RSA (residenze per anziani) dove un cluster di influenza/RSV può avere conseguenze serie – poter testare immediatamente in situ tutti i pazienti sintomatici consente di isolare i positivi e proteggere gli altri ospiti con rapidità.
Va sottolineato che la facilità d’uso di queste soluzioni ne amplia i contesti applicativi: operatori sanitari con formazione di base (es. infermieri, tecnici) possono eseguire i test grazie a interfacce guidate e kit preassemblati. Questo è fondamentale in ambienti come cliniche da campo, postazioni di frontiera o reparti a basso personale: la tecnologia è progettata per inserirsi senza attriti nel flusso di lavoro.
7. Impatto clinico e operativo
L’introduzione della PCR multiplex decentralizzata in contesti clinici ha mostrato benefici tangibili misurabili. Il primo indicatore è la drastica riduzione del tempo di refertazione (TAT). Meta-analisi su studi ospedalieri indicano che l’uso di test “sample-to-answer” rapidi riduce i tempi di risposta di circa 24 ore rispetto ai test PCR tradizionali inviati al laboratorio [1]. In pratica, un risultato che con il consueto percorso arriverebbe il giorno seguente (mediamente >24h), con la PCR point-of-care viene ottenuto in meno di 2 ore dall’esecuzione del campione. Questo delta di un giorno intero è cruciale nel modificare gli esiti clinici. Ad esempio, uno studio ha riscontrato una riduzione della degenza media di ~0,8 giorni per i pazienti gestiti con diagnostica molecolare rapida rispetto a quelli con iter standard [1]. Dimettere quasi un giorno prima un paziente significa liberare posti letto, ridurre il rischio di infezioni nosocomiali aggiuntive e tagliare i costi ospedalieri.
Un altro impatto significativo è sulla appropriatezza terapeutica. Nel caso dell’influenza, disporre del risultato in real-time aumenta del 25-30% la probabilità che i pazienti positivi ricevano subito la terapia antivirale (es. oseltamivir) appropriata [19]. Contestualmente, si è osservato un aumento fino al 55% nell’adozione tempestiva di misure di isolamento e controllo delle infezioni (stanze singole, coorte influenza) per i pazienti diagnosticati rapidamente [20]. Ciò contribuisce a contenere la diffusione nosocomiale dei virus e proteggere pazienti e operatori.
Dal lato della stewardship antibiotica, i dati suggeriscono che la disponibilità di una diagnosi virologica rapida porti a una riduzione dell’uso empirico di antibiotici in pazienti con infezioni virali confermate. In generale, identificare un virus come causa di sintomi respiratori permette ai clinici di sospendere o evitare gli antibiotici in quei casi, focalizzandoli solo sui pazienti con evidenza di infezione batterica. Le linee guida di gestione antimicrobica sottolineano che i test diagnostici rapidi dovrebbero essere integrati nei programmi di stewardship per ridurre l’uso inappropriato di antibiotici [21]. Sebbene gli studi sugli adulti abbiano dato risultati talora eterogenei, diverse ricerche mostrano riduzioni (anche se moderate) nell’impiego di antibiotici e nella durata della terapia quando i risultati virali vengono comunicati rapidamente al medico [22]. Un esempio pratico: in un ospedale, passando da un pannello PCR tradizionale con TAT ~27 ore a uno rapido da 1-2 ore, si è osservata una diminuzione significativa delle prescrizioni di antibiotici empirici nelle prime 24 ore di ricovero [23]. Ciò indica che i medici, disponendo di un test molecolare rapido, sono più propensi ad aspettare l’esito per indirizzare la terapia invece di partire “al buio” con coperture antibatteriche ampie.
Un ulteriore beneficio operativo è la riduzione di indagini diagnostiche aggiuntive. Sapere subito che un paziente ha, ad esempio, influenza A, può evitare una serie di altri esami (radiografie, marker infiammatori ripetuti, consulenze) che spesso vengono richiesti nel tentativo di chiarire una diagnosi incerta. Uno studio pediatrico ha evidenziato che i bambini risultati positivi per influenza tramite test rapido hanno subito meno esami invasivi e di laboratorio rispetto a quelli con diagnosi negativa o incerta [24], segno che una diagnosi molecolare chiara all’inizio del percorso può snellire il workup clinico.
Dal punto di vista epidemiologico e di sanità pubblica, la diagnostica decentralizzata offre infine un vantaggio di sorveglianza attiva. Raccogliendo in cloud i risultati in tempo reale da varie sedi, si può creare un sistema di allerta precoce per focolai: se in vari pronto soccorso periferici compare un aumento insolito di casi RSV positivi, il laboratorio centrale o le autorità possono venirne a conoscenza immediatamente e reagire (es. allertare reparti pediatrici, verificare coperture vaccinali, etc.). Questo concetto di network diagnostico distribuito e interconnesso trasforma ogni punto di cura in una sentinella epidemiologica, senza attendere i consolidati tempi di notifica dei laboratori centralizzati.
In sintesi, i dati e l’esperienza sul campo confermano che la Real-Time PCR decentralizzata non è solo un vezzo tecnologico, ma si traduce in outcome migliori: diagnosi più veloci -> terapie più mirate -> degenze più brevi -> meno costi e meno resistenze. Una win-win sia per i pazienti che per il sistema sanitario.
«L’implementazione diffusa di test PCR multiplex rapidi e decentralizzati innalza il livello della risposta clinica. In contesti ad alto flusso come il pronto soccorso, passare da un referto in 24-48 ore a un referto in 90-120 minuti cambia radicalmente le decisioni: significa poter isolare subito un paziente infetto, iniziare terapie mirate durante il triage e, soprattutto, evitare antibiotici inutili quando si identifica un virus. Dal punto di vista tecnologico, siamo riusciti a portare la potenza di un laboratorio centralizzato direttamente al letto del paziente, con risultati immediatamente disponibili anche al centro per la sorveglianza. Questa convergenza tra hardware portatile e software cloud accorcia le distanze in sanità: il risultato giusto, nel posto giusto, al momento giusto.»
— Lorenzo Colombo, CTO di Ulisse Biomed
Conclusioni
La diagnostica molecolare decentralizzata delle patologie respiratorie rappresenta oggi una realtà concreta, frutto della convergenza tra innovazione tecnologica e necessità cliniche pressanti. Abbiamo visto come le piattaforme di Real-Time PCR multiplex portatili, supportate da ecosistemi cloud e algoritmi di intelligenza artificiale, stiano ridefinendo i tempi e i modi della diagnosi in contesti ospedalieri e territoriali. I benefici sono molteplici e sinergici: riduzione drastica del TAT, maggiore appropriatezza nell’uso di antivirali e antibiotici, miglioramento delle misure di controllo delle infezioni e, in ultima analisi, outcome clinici migliori per i pazienti. Inoltre, questo modello distribuito crea una rete di dati in tempo reale che può potenziare la sorveglianza epidemiologica e la risposta a focolai emergenti, colmando il gap tra territorio e centro.
Dal punto di vista strategico, investire nella decentralizzazione della diagnostica significa rendere il sistema sanitario più reattivo e resiliente. Un pronto soccorso dotato di PCR point-of-care è in grado di affrontare con efficacia sia la comune influenza stagionale sia nuove minacce pandemiche, senza attendere conferme esterne. Allo stesso tempo, la centralizzazione dei dati tramite piattaforme cloud consente a laboratori e autorità sanitarie di mantenere il controllo di qualità e la visione d’insieme, assicurando che questa democratizzazione della diagnostica avvenga in modo sicuro e armonizzato.
Certo, vi sono ancora sfide e margini di miglioramento: dai costi per test ancora elevati rispetto ai test antigenici, alla necessità di formare il personale all’uso corretto di queste tecnologie, fino alla piena integrazione nei percorsi clinici e organizzativi esistenti. Ma il trend è tracciato: come confermano le analisi di mercato, il segmento della diagnostica molecolare decentralizzata (e in particolare della PCR portatile) è in crescita a doppia cifra e destinato a diventare uno standard di cura entro il prossimo decennio. Organizzazioni autorevoli e letteratura scientifica iniziano a riconoscere apertamente che “routine mPOCT should replace laboratory-based diagnostics” almeno in determinati contesti e stagioni.
In conclusione, la Real-Time PCR decentralizzata per infezioni respiratorie non è più solo una visione futuristica, ma una soluzione attuale che sta rivoluzionando la diagnostica rapida. Laboratori ospedalieri, cliniche e pronto soccorsi hanno oggi la possibilità di abbracciare questo cambio di paradigma, con benefici clinici ed economici comprovati. La sfida ora è di scalare queste innovazioni, integrarle nei protocolli standard e garantirne la sostenibilità economica, affinché ogni paziente – dovunque si trovi – possa ottenere la diagnosi giusta al momento giusto.
Fonti e bibliografia
[1] [19] [20] Routine Rapid Multiplex PCR for Respiratory Viruses Improves Inpatient Outcomes - Pulmonology Advisor
[2] [10] [11] [12] [13] Rapid Influenza Diagnostic Tests | Influenza (Flu) | CDC
https://www.cdc.gov/flu/hcp/testing-methods/clinician_guidance_ridt.html
[3] [21] [22] [28] Multiplex Respiratory Virus Testing for Antimicrobial Stewardship: A Prospective Assessment of Antimicrobial Use and Clinical Outcomes Among Hospitalized Adults - PMC
https://pmc.ncbi.nlm.nih.gov/articles/PMC5853820/
[4] [5] [6] [7] [8] [15] [16] [17] [18] ID15 - The Ulisse Biomed Ecosystem: the Hyris System™ modular diagnostic platform
[9] Influenza and Bacterial Coinfection in Adults With Community ...
https://academic.oup.com/ofid/article/7/3/ofaa066/5760732
[14] Key Insights into Respiratory Virus Testing: Sensitivity and Clinical ...
https://www.mdpi.com/2076-2607/13/1/63
[23] Impact of multiplex molecular assay turn-around-time on antibiotic ...
https://www.sciencedirect.com/science/article/abs/pii/S138665321830283X
[24] Rapid Influenza Diagnosis in Children With Serious Bacterial ...
https://onlinelibrary.wiley.com/doi/10.1111/apa.70174
[25] Clinical impact of a routine, molecular, point-of-care, test-and-treat strategy for influenza in adults admitted to hospital (FluPOC): a multicentre, open-label, randomised controlled trial - PubMed
https://pubmed.ncbi.nlm.nih.gov/33285143/
[26] [27] Rapid point of care diagnostic tests for viral and bacterial respiratory tract infections--needs, advances, and future prospects - PubMed